En ese momento me dio la luz para escribir este post (no sabia sobre que escribir, con tantas cosas que se pueden hacer!), ya que la entrada de datos en R, es sumamente importante y teniendo claro esto, muchas de las cosas que uno hará en R serán "breves*" (como decimos los "ñeros" jejeje).
*BREVE= en el lenguaje "ñeristico", significa, fácil, sin complique, suave, etc!
Para empezar, aclarare donde deben estar nuestros archivos de datos en nuestro computador y como decirle a R, donde estan o como decirle cuando cambiar de lugar para trabajar.
Entonces, como bien sabemos en nuestro computador podriamos tener 3.548.125 carpetas en lugares como "C", "D", el Escritorio u otros dentro de estos o afuera de estos, por ejemplo:
Podría tener un archivo en formato ".txt" (mas adelante explicare lo de los formatos!) , llamado "arc_1.txt" en mi escritorio:
O podria tener otro archivo llamado "arc_2.txt" en una carpeta personal, en este caso (una carpeta mia llamada "morphometria"):
Entonces, como puedo tener archivos en muchas carpetas (no solo en estas dos), debo indicarle a R donde se encuentran los archivos que voy a utilizar en esta oportunidad; esto lo podemos hacer via linea de comandos, o lo podemos hacer graficamente.
En windows es muy sencillo hacerlo, puesto que solo se debe dar click, en "cambiar el directorio".
En linux es aun mas sencillo porque solo se debe utilizar un comando con la ruta que estemos trabajando, este comando es "setwd".
Por ejemplo, si quiero decirle que vamos a trabajar sobre el escritorio en donde esta mi archivo "arc_1.txt", le damos:
> setwd ("/home/ambrosio/Escritorio")
Y si por ejemplo, queremos trabajar en mi carpeta "morphometria", donde tengo mi archivo "arc_2.txt", solo le tenemos que indicar la ruta para acceder a la carpeta:
> setwd ("/home/ambrosio/morphometria")
Y listo!!!, de este modo le indicamos a R, donde están nuestros archivos!!
Muchas veces, a muchas personas se les hace difícil, saber la ruta, porque es una ruta muy larga o porque no la conocen, para esto solo deben pararse sobre el archivo que tenga sus datos, le dan click derecho, después le damos click a propiedades y aparecerá la ruta del archivo:
Una vez estemos en el directorio sobre el que queramos trabajar, tenemos que incluir los archivos de datos que queramos utilizar dentro de el directorio (aunque en el mismo R podemos escribir los datos con los que vamos a trabajar, como vectores, tablas, etc etc etc. pero este no es el objeto de este post, ya que estamos trabajando acerca incluir datos a R).
Para esto lo primero que tenemos que tener en cuenta son los formatos de los archivos, personalmente los archivos que yo utilizo, estan en formato .csv o .txt, es decir archivos separados por comas "," o archivos separados por espacios " ".
son muy faciles de utilizar porque pueden ser modificados en excel (para los que aman excel!!) o en editores de texto como Geany (para los que amamos geany!!) y es sencillo manejar grandes cantidades de datos con estos formatos!!!
Para aprender a crear archivos .csv y .txt he generado unos datos !IMAGINARIOS¡ acerca de algunas variables "medidas" en las 7 etapas del barrio Zapamanga de Floridablanca/Santander. Los datos fueron incluidos dentro de una tabla utilizando LibreOffice 3.3 (Abajo excel!), de allí vamos a guardarlos en formato .csv y en geany los transformaremos en formato .txt (para observar como es de fácil transformar en geany formatos sin tener que ir de nuevo a hojas de calculo y guardar en formato .txt .
Los datos originales en una tabla son estos:
Al abrirlo en geany se vería así y no se distinguirán los decimales:
Entonces lo único que tenemos que hacer es seleccionar y copiar los datos de la hoja de calculo y pegarlos en una hoja en blanco de geany (o con el editor de texto que estemos trabajando), nos vamos a la solapa que dice "Buscar" damos clic ahi, y despues damos clic en "Reemplazar", finalmente le decimos que reemplace las comas por puntos en el documento y ya tenemos nuestros decimales definidos por puntos (ahora lo que tenemos que hacer es separar nuestras columnas por comas o por espacios, segun lo que queramos hacer (o .txt o .csv).
Para separar nuestras columnas entonces seleccionamos un espacio en blanco entre dos números:
Si hubiésemos querido el archivo en formato .txt lo unico que teniamos que hacer era reemplazar la seleccion hecha en el paso anterior por un espacio en blanco y ya!!!.
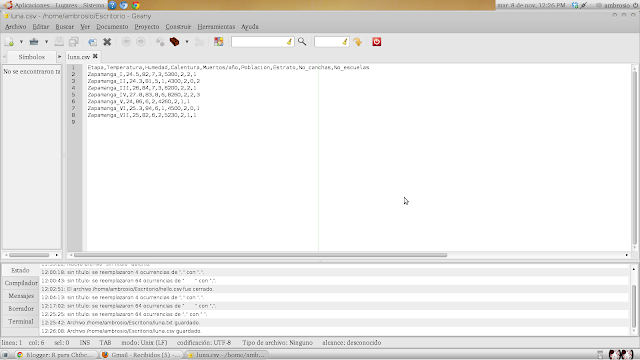
Obviamente una vez terminada nuestra edición debemos guardar el archivo en la carpeta que estemos trabajando, entonces, nos vamos en geany a "Archivo", le damos "Guardar como" y nombramos el archivo como "luna.csv" (en honor a la nueva mascota de Juancho) y sí la tenemos en formato .txt la nombramos "luna.txt". Este seria el aspecto final de los archivos en ambos formatos:
Archivo en formato .txt (luna.txt)
Archivo en formato .csv (luna.csv)
Una vez teniendo listos nuestros datos en la carpeta que estemos utilizando, los incluiremos en R, para esto necesitamos simples comandos de lectura en R, dependiendo si el formato que vamos a leer es .csv o .txt.
empezaremos llamando nuestro formato .csv
> chorizo <- read.csv("luna.csv",header=T)
#Este comando indica que crearemos un objeto con el nombre "chorizo" en R, a partir de una matriz de datos externa (nuestro "luna.csv"), para esto se utiliza la función "read.csv" y lo que indica que el nuevo objeto en R se llamara chorizo es la flecha "<-", el argumento header=T o header=TRUE, significa que la primer linea o fila de la matriz, son los nombres de las variables, de nos ser así, se tendría que poner, header=F o header=FALSE.
Para ver el nuevo objeto que hemos creado en R, solo tenemos que darle el nombre del objeto es decir "chorizo"
> chorizo
# Y veremos algo así en la ventana de R:
Para ver el nuevo objeto que hemos creado en R, solo tenemos que darle el nombre del objeto es decir "chorizo"
> chorizo
# Y veremos algo así en la ventana de R:
Etapa Temperatura Humedad Calentura Muertos.año Poblacion Estrato
1 Zapamanga_I 24.5 82 7 3 5300 2
2 Zapamanga_II 24.3 81 5 1 4300 2
3 Zapamanga_III 26.0 84 7 3 6200 2
4 Zapamanga_IV 27.8 83 8 6 8260 2
5 Zapamanga_V 24.0 86 6 2 4260 2
6 Zapamanga_VI 25.3 84 6 1 4500 2
7 Zapamanga_VII 25.0 82 6 2 5230 2
No_canchas No_escuelas
1 2 1
2 0 2
3 2 1
4 2 3
5 1 1
6 0 1
7 1 1
Después leeremos esta misma matriz de datos pero en formato .txt y utilizaremos el siguiente comando:
> galletas <- read.table("luna.txt",header=T)
#Con este comando indicamos que queremos crear un objeto en R con el nombre de "galletas", para eso utilizamos la función read.table (que es para leer archivos en formato.txt, el argumento "header=T", se utiliza de la misma forma como con la función read.csv, lo mismo que el uso de la flecha.
para ver el objeto hacemos lo mismo que hicimos antes, pero en vez de escribir en R "chorizo", escribimos "galletas" y veremos nuestros datos.
> galletas
#asi veremos los mismos datos que mostré anteriormente.
Una vez teniendo nuestros objetos creados en R, vamos a ver un poco acerca de como manejarlos y un tanto "jugar con ellos".
Primero que todo, debemos saber (si queremos!) que objetos hemos creado en R, para esto solo le damos el comando:
>objects ()
#esto nos muestra los objetos que hemos creado en R:
[1] "chorizo" "galletas"
En ocasiones necesitamos hacer analisis con solo algunas variables de las matrices, o solo necesitamos tomar algunos datos de esta; para eso podemos utilizar submatrices, que son muy facil de crear, o se pueden tomar los datos directamente an el analisis, utilizando los mismos argumentos de creacion de submatrices.
Por ejemplo si queremos tomar solo las variables temperatura y humedad, le indicamos con un comando que esas variables corresponden a las columnas 2 y 3 y el damos el comando:
> choricito<-(chorizo[,2:3])
#con esto le decimos que tome de la matriz original "chorizo" las columnas 2 y 3 y cree una submatriz llamada "choricito", asi que al darle el siguiente comando, nos aparecera la nueva mariz:
>choricito
Temperatura Humedad
1 24.5 82
2 24.3 81
3 26.0 84
4 27.8 83
5 24.0 86
6 25.3 84
7 25.0 82
Y si queremos decirle que tome las 4 primeras variables solo tenemos que darle el sigueinte comando:
>a<-(chorizo[,1:4])
>a
Etapa Temperatura Humedad Calentura
1 Zapamanga_I 24.5 82 7
2 Zapamanga_II 24.3 81 5
3 Zapamanga_III 26.0 84 7
4 Zapamanga_IV 27.8 83 8
5 Zapamanga_V 24.0 86 6
6 Zapamanga_VI 25.3 84 6
7 Zapamanga_VII 25.0 82 6
Ahora bien, esto lo realizamos para tomar el numero de columnas deseado o las variables que queramos incluir en el análisis, pero si por ejemplo queremos mas bien escoger el numero de filas o el numero de lugares a evaluar lo único que tenemos que hacer es cambiar la posición de la coma en el comando y ponerla al final, es decir:
>alto<-(chorizo[1:4,])
>alto
Etapa Temperatura Humedad Calentura Muertos.año Poblacion Estrato
1 Zapamanga_I 24.5 82 7 3 5300 2
2 Zapamanga_II 24.3 81 5 1 4300 2
3 Zapamanga_III 26.0 84 7 3 6200 2
4 Zapamanga_IV 27.8 83 8 6 8260 2
No_canchas No_escuelas
1 2 1
2 0 2
3 2 1
4 2 3
> galle<-(luna[2:4,])
>galle
> galle<-(luna[2:4,])
>galle
Etapa Temperatura Humedad Calentura Muertos.año Poblacion Estrato
2 Zapamanga_II 24.3 81 5 1 4300 2
3 Zapamanga_III 26.0 84 7 3 6200 2
4 Zapamanga_IV 27.8 83 8 6 8260 2
No_canchas No_escuelas
2 0 2
3 2 1
4 2 3
Con esta pequeña introduccion al manejo de datos en R, es muy facil empezar a hacer nuestros analisis, otra herramienta muy util en el manejo de datos son los archivos multiples tipo "Array", que explique en uno de mis post anteriores:
Array's en R
Espero que este post, haya sido de gran utilidad, especialmente para las personas que estan empezando a utilizar R.












Excelente amigo..."mas claro no canta un gallo"..gracias.
ResponderEliminarBacan Gracias, quedo bueno, es decir concreto. Estaré atento para cuando postee los trucos de R, y por supuesto, Los trucos de trucos. ..y que viva CHIBCHOMBIA.
ResponderEliminarOk.. facil,"breve"
ResponderEliminarGracias me hiciste un parote :D
ResponderEliminarGracias!! Tengo una pregunta, espero alguien me pueda guiar: Se puede hacer un scatter plot en R con solo algunas columnas de la matriz?, es para hacer el grafico por partes puesto que al ser muchas variables el grafico ya no se ve bien, y no se puede apreciar alguna relacion entre variables.
ResponderEliminarhola buenos dias, espero me puedan hechar una mano, estoy haciendo un trabajo en la consola R, pero no encuentro la manera de que R, lea el archivo que prepare en EXCEL con extension CSV,me pòdrian indicar paso a paso que es lo que debo hacer para que R lea esta data????, he seguido los pasos que ustedes han indicado en el blog paso por paso sin embargo la consola me da error.... estoy desesperada pues tengo que enviar esta tarea hasta el sabado... gracias
ResponderEliminarHola...gracias por tu comentario en el blog!!
ResponderEliminarpuedes también intentar importar la matriz utilizando el paquete RODBC
>install.packages("RODBC")
>library(RODBC)
puedes seguir la rutina que indico en el ultimo post!!!:
http://rchibchombia.blogspot.com/2012/12/introduccion-al-programa-estadistico-r.html
específicamente en las diapositivas del día numero 1:
https://docs.google.com/file/d/0B9ogQEXTywHsT29UMnhfcXVtX28/edit
en ese ultimo post extendí muchos de los temas que he manejado en el blog, por lo cual te podría ser de mucha ayuda!!
si por alguna razón no te funciona la rutina ni el paquete..
me envías tu matriz de datos por correo (atorresgalvis@gmail.com) y yo le pego una revisada rápida para ver si le encuentro algún error de construcción!!
"que la fueRza te acompañe!!"
Broxi y si yo quiero que R me cree una submatriz únicamente con columnas que no estén juntas, por ejemplo: la columna 2 y la 9?
ResponderEliminarufff buenísimo, gracias por el post!
ResponderEliminarQuiero que me indiquen cómo se puede guardar los trabajos en R ya estoy cansado y es que no me sale.
ResponderEliminarSoy Manuel desde Madrid por favor que me indiquen.y por otra parte cómo se importa los datos de excel a R.
hoy aprendi algo de R, gracias, continuare en la lucha
ResponderEliminarno soy capaz de abrir un texto en txt que tengo dentro de una carpeta de mi escritorio. C//Documents and setting/usuario/Escritorio/sinopses txt/2008.txt, y el proceso que sigo es:
ResponderEliminar-Abrir R
-cambiar el directorio a esta carpeta
-descargar el documento
-instalar librería (tm)
y colocar el comando: txt<-readLines ("2008.txt", encoding=UTF-8")
Tras esto aparece el error "not open file (con, "r") not found file or directory
Que puede ser?
Además me gustaría crear un corpus con varios txt, son sinopsis de novelas sobre el camino de Santiago publicadas entre 2008 y 2012 haciendo un txt para cada año con título, fecha y sinopses, sabeis como puedo reunirlas. Gracias. Lo he visto con twitts pero non con textos!
Muchas gracias por este post, me sacaste de un apuro.
ResponderEliminarEste comentario ha sido eliminado por un administrador del blog.
ResponderEliminarhola. tengo el siguiente vector de datos x<- c(3024191518), pero quiero darle orden a R que los separe por comas de tal forma que los pueda leer asi: c(30, 24, 19, 15, 18)y poder calcular por ejemplo su media. Como seria la funcion u orden para que me incorpore las comas y espacios.
ResponderEliminarGracias
Lo primero que tienes que hacer es saber que no estás introduciendo un vector de datos sino un vector de DATO (sin la "s") ya que estás poniendo un vector que contiene un solo dato y R por defecto lo lee como un vector numérico que contiene un solo valor.
Eliminar> x <- c(3024191518) #Como tu estás tratando de crearlo
> length(x) #Para ver cual es la longitud del vector que acabas de crear (será 1!)
> class(x) #Para ver que tipo de vector acabas de crear (es Númerico)
De manera que lo primero que debes hacer es crear el vector pero indicándole que es un vector de caracteres para después poder cortar la frase:
> x<- as.character(c(3024191518)) #Creas el vector de un solo caracter
> x2 <- as.numeric(substring(x, seq(1,nchar(x),2), seq(2,nchar(x),2)))
# Lo cortas cada 2 posiciones y le das un nombre al nuevo vector "X2", éste si es de datoS
> length(x2)
> class(x2)
Y ya utilizando el vector x2 puedes hacer tus cálculos:
> mean(x2) #Calculas la media de tu vector de DATOS
#[nota: si no te importa perder el vector original "x" puedes ponerle el nombre "x" cuando lo cortes en vez de "x2".]
SALUDOS!
Este es un ejercicio? Dónde puedo descargar la tabla para hacerlo? Es la primera vez que usaré R, soy demasiado noob en esto. Cualquier ayuda es de mucha utilidad.
ResponderEliminarHola, más que ejercicio es como una pequeña explicación.
EliminarSin embargo si puedes intentar hacerlo como ejercicio, solo que no tengo el archivo original, puedes intentarlo con alguna tabla que tengas o generas una tabla dummy en excel para que lo intentes.
Muchas gracias por el interés en el blog y perdón por la demora para contestar.
Saludos
Saludos Alguien me puede ayudar si hay alguna función para reducir decimales de los números de los ejes de coordenadas cuando realizo una dispersión de datos en plot, un eje me sale con 2 decimales y el otro eje me sale con 3 decimales.Quiero dejar con dos Decimales a las ejes "x" y "y". Además no se como hacer que la numeración empiece desde el punto de unión, hasta el final de los ejes "x" y "y". Osea en otras palabras no tenga porcentaje de margenes sino que la escala este desde el comienzo hasta el final tanto para el eje x e y. Gracias Espero que alguien me pueda ayudar con un ejemplo claro.
ResponderEliminarFUNCIÓN PARA REDUCIR EL NUMERO DE "DECIMALES EN EJES COORDENADOS" CUANDO SE REALIZA UNA GRÁFICA DE DISPERSIÓN DE DATOS MEDIANTE LA FUNCIÓN PLOT. AGRADEZCO ANTICIPADAMENTE AQUELLA PERSONA AMABLE QUE ME PUEDA AYUDAR. COMO PUEDO REDUCIR PARA DEJAR A UNA ESCALA. LO AGRADEZCO MUCHO A LA PERSONA MUY AMABLE.
ResponderEliminarPor favor, podria explicar esto? se lo agradezco....
ResponderEliminartengo que hacer una function para trabajar con las columnas de un file csv. los files estan en un directorio estos son 300 y pico y se llamam 001...300.csv. por los argumentos de la function le doy el directorio, la columa de la cual deseo la media y una lista tipo rango de que files quiero utilizar. el problema al tratar de hacer la function con read.csv, es que no reconoce el file alli dentro
> setwd("C:/Users/Vilma Contreras/Desktop/Vilma2017/R")
> pollutantmean <- function(directory,pollutant, id=1:332){
+ files <- list.files(path = "C:/Users/Vilma Contreras/Desktop/Vilma2017/R/directory")
+ for (i in id) {
+ con<-file(files[i],"r")
+
+ tmp<-read.csv(con)
+ data<-rbind(data,tmp)
+ }
+ }
> pollutantmean("specdata","sulfate",1:2)
Error in file(files[i], "r") : cannot open the connection
In addition: Warning message:
In file(files[i], "r") : cannot open file 'NA': No such file or directory
...si lo fuera de la function si trabaja, pero no dentro de la function
mydata<-read.csv("001.csv",header=T)
mean(mydata$sulfate,na.rm=TRUE)
## [1] 3.880701
Hola!
EliminarMe parece que tienes un problema de variables locales y variables globales.
Es un tipico problema cuando trabajas con list.files o con Sys.glob.
Lo que yo te recomendaria es que hicieras la lista de archivos list.files por fuera de la función y ahí si la ejecturás, al final no es que pierdas mucha funcionalidad haciendo por fuera la lista de archivos.
Por otra parte, no entiendo muy bien lo que quieres que haga tu función, sin embargo te dejo un ejemplo de como yo lo haría si es que necesitará resolver el problema con prontitud:
1. Construyo la función:
pollutantmean <- function(pollutant, desde = 1, hasta = 2){
datos <- NULL
for (i in desde:hasta) {
tmp <- read.csv(archivos[i])
toDatos <- tmp[pollutant][,1]
datos <- c(datos, toDatos)
}
mean(datos)
}
2. Este paso es opcional pues depende de si estoy en el directorio que deseo o no:
#Escojo el nombre del directorio al que deseo ir ADENTRO de en el que estoy
directory <- "directorio"
#Me voy a trabajar al directorio que deseo
setwd(paste(getwd(), "/", directory, sep=""))
3. Hago la lista de archivos iniciales:
archivos <- list.files()
4. Corro la función:
pollutantmean("sulfate",desde= 20, hasta= 25)
EliminarRecuerda que debes tener ordenados tus files para poderlos leer con read.csv, ya que como escribí la función lo está leyendo teniendo en cuenta el orden de los archivos en la lista de archivos creada con "archivos[i]", de tal modo que si quisieras llamarlos tomando el nombre de los archivos tendría que ser de otro modo. Pero en tu caso no importa, ya que los nombres del los archivos son "001", "002", etc. De manera que al crear la lista de archivos lo hace de forma ordenada numérica.
Espero te haya sido útil la respuesta y te ayude a resolver tu problema.
SALUDOS
Muy chistoso y muy útil. Me encantó y soy chibchombiana
ResponderEliminarque bueno que te haya gustado te haya gustado. Saludos!
Eliminarbuenas tengo una pequeña duda para hacer un boxplot aplicado a un modelo de regresión simple, mi variable "y" es una variable continua y mi "x" es una variable que contiene cuatro factores y doy el comando > boxplot(y ~ x) y da lo siguiente Error in boxplot.default(split(mf[[response]], mf[-response]), ...) :
ResponderEliminaradding class "factor" to an invalid object